Erfahren Sie, wie Sie OCR-Tools, Apache Spark und andere Apache Hadoop-Komponenten verwenden, um PDF-Bilder in großem Umfang zu verarbeiten.
Technologien zur optischen Zeichenerkennung (OCR) haben sich in den letzten 20 Jahren erheblich weiterentwickelt. In dieser Zeit gab es jedoch wenig oder keine Bemühungen, OCR mit verteilten Architekturen wie Apache Hadoop zu verbinden, um eine große Anzahl von Bildern nahezu in Echtzeit zu verarbeiten.
In diesem Beitrag erfahren Sie, wie Sie Standard-Open-Source-Tools zusammen mit Hadoop-Komponenten wie Apache Spark, Apache Solr und Apache HBase verwenden, um genau das für einen Anwendungsfall für Informationen zu medizinischen Geräten zu tun. Insbesondere werden Sie einen öffentlichen Datensatz verwenden, um narrativen Text in durchsuchbare Felder umzuwandeln.
Obwohl sich dieses Beispiel auf Informationen zu medizinischen Geräten konzentriert, kann es in vielen anderen Szenarien angewendet werden, in denen die Verarbeitung und Speicherung von Bildern erforderlich ist. Versicherungsunternehmen können beispielsweise alle ihre gescannten Dokumente in Schadenakten durchsuchbar machen, um eine bessere Schadenbearbeitung zu ermöglichen. In ähnlicher Weise könnte die Lieferkettenabteilung in einer Produktionsstätte alle technischen Datenblätter von Teilelieferanten scannen und sie für Analysten durchsuchbar machen.
Anwendungsfall:Registrierung von Medizinprodukten
In den letzten Jahren hat sich im Bereich der elektronischen Registrierung von Arzneimitteln eine Flut von Änderungen ergeben. Der ISO-Standard IDMP (Identification of medical products) ist ein solches Nachrichtenformat zur Registrierung von Produkten und den darin enthaltenen Substanzen, wobei die Arzneimittel-ID, Verpackungs-ID und Chargen-ID verwendet werden, um die Produkte im Falle unerwünschter Erfahrungen zu verfolgen, illegal Import, Fälschung und andere Fragen der Pharmakovigilanz. Der Standard fordert, dass nicht nur neue Produkte registriert werden müssen, sondern dass auch die älteren/archivierten Unterlagen aller Produkte, denen die Öffentlichkeit ausgesetzt sein könnte, in elektronischer Form bereitgestellt werden müssen.
Um die IDMP-Standards in verschiedenen Unternehmen einzuhalten, müssen Unternehmen in der Lage sein, Daten aus mehreren Datenquellen wie RDBMS sowie in einigen Fällen aus älteren Produktdatenblättern abzurufen und zu verarbeiten. Während es bekannt ist, Daten aus RDBMS über Technologien wie Apache Sqoop aufzunehmen, erfordert die Verarbeitung von Legacy-Dokumenten etwas mehr Arbeit. Zum größten Teil müssen die Dokumente aufgenommen werden und relevanter Text muss programmgesteuert in großem Umfang unter Verwendung vorhandener OCR-Technologien extrahiert werden.
Datensatz
Wir verwenden einen Datensatz der FDA, der alle 510(k)-Anmeldungen enthält, die seit 1976 jemals von Herstellern medizinischer Geräte eingereicht wurden. Abschnitt 510(k) des Food, Drug and Cosmetic Act verlangt von Geräteherstellern, die sich registrieren müssen, eine Benachrichtigung FDA über ihre Absicht, ein medizinisches Gerät auf den Markt zu bringen, mindestens 90 Tage im Voraus.
Dieser Datensatz ist in diesem Fall aus mehreren Gründen nützlich:
- Die Daten sind kostenlos und gemeinfrei.
- Die Daten passen genau zur europäischen Verordnung, die im Juli 2016 in Kraft tritt (wo Hersteller neue Datenstandards einhalten müssen). FDA-Füllungen enthalten wichtige Informationen, die für die Ableitung eines vollständigen Überblicks über IDMP relevant sind.
- Das Format der Dokumente (PDF) ermöglicht es uns, einfache, aber effektive OCR-Techniken beim Umgang mit Dokumenten in mehreren Formaten zu demonstrieren.
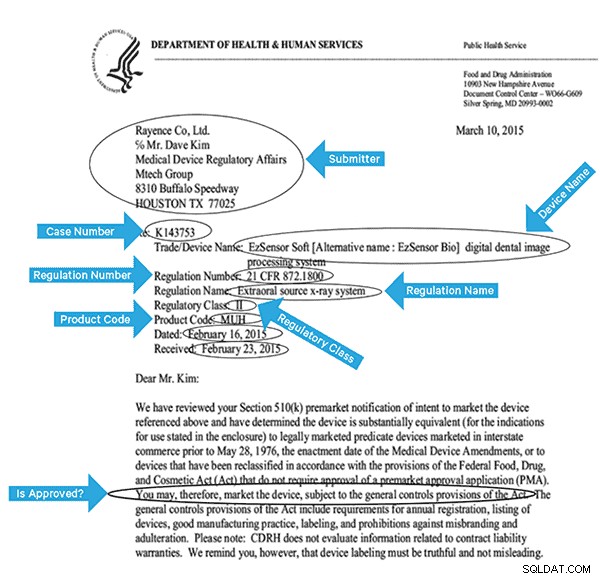
Um diese Daten effektiv zu indizieren, müssen wir einige Felder aus den Bildern extrahieren. Nachfolgend finden Sie ein Beispieldokument mit den möglichen Feldern, die extrahiert werden können.
High-Level-Architektur
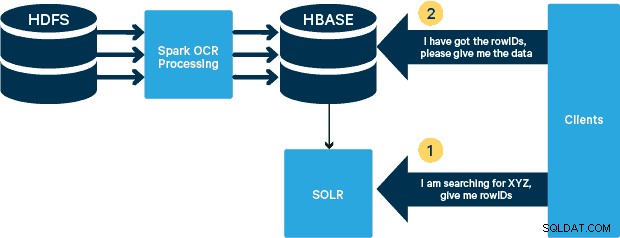
Für diesen Anwendungsfall werden die PDFs in HDFS gespeichert und mit Spark- und OCR-Bibliotheken verarbeitet. (Der Erfassungsschritt würde den Rahmen dieses Beitrags sprengen, könnte aber so einfach sein wie das Ausführen von hdfs -dfs -put oder über eine webhdfs-Schnittstelle.) Spark ermöglicht die Verwendung von nahezu identischem Code in einer Spark-Streaming-Anwendung für nahezu Echtzeit-Streaming, und HBase ist ein perfektes Speichermedium für wahlfreien Zugriff mit geringer Latenz – und eignet sich gut zum Speichern von Bildern mit die neue MOB-Funktionalität, um zu booten. Cloudera Search (das auf Apache Solr aufbaut) ist die einzige Suchlösung, die sich nativ in HBase integrieren lässt, sodass Sie sekundäre Indizes erstellen können.
Einrichten der Medizinproduktetabelle in HBase
Wir werden das Schema für unseren Anwendungsfall einfach halten. Die rowID ist der Dateiname, und es gibt zwei Spaltenfamilien:„info“ und „obj“. Die Spaltenfamilie „Info“ enthält alle Felder, die wir aus den Bildern extrahiert haben. Die Spaltenfamilie „obj“ enthält die Bytes des eigentlichen binären Objekts, in diesem Fall PDF. Der Name der Tabelle lautet in unserem Fall „mdds.“
Wir werden die in HBASE-11339 eingeführte HBase-MOB-Funktionalität (Medium Object) nutzen. Um HBase für die Verarbeitung von MOB einzurichten, sind einige zusätzliche Schritte erforderlich, aber praktische Anweisungen finden Sie unter diesem Link.
Es gibt viele Möglichkeiten, die Tabelle programmgesteuert in HBase zu erstellen (Java-API, REST-API oder eine ähnliche Methode). Hier verwenden wir die HBase-Shell, um die „mdds“-Tabelle zu erstellen (wobei wir absichtlich einen aussagekräftigen Spaltenfamiliennamen verwenden, um das Nachvollziehen zu erleichtern). Wir möchten, dass die „info“-Spaltenfamilie nach Solr repliziert wird, aber nicht die MOB-Daten.
Der folgende Befehl erstellt die Tabelle und aktiviert die Replikation für eine Spaltenfamilie namens „info“. Entscheidend ist die Angabe der Option REPLICATION_SCOPE => '1' , sonst erhält der HBase Lily Indexer keine Updates von HBase. Wir möchten den MOB-Pfad in HBase für Objekte verwenden, die größer als 10 MB sind. Um dies zu erreichen, erstellen wir auch eine weitere Spaltenfamilie namens „obj“ mit den folgenden Parametern für MOBs:
IS_MOB => wahr, MOB_THRESHOLD => 10240000
Der IS_MOB Der Parameter gibt an, ob diese Spaltenfamilie MOBs speichern kann, während MOB_THRESHOLD gibt danach an, wie groß das Objekt sein muss, damit es als MOB betrachtet wird. Lassen Sie uns also die Tabelle erstellen:
erstelle 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000} Um zu bestätigen, dass die Tabelle ordnungsgemäß erstellt wurde, führen Sie den folgenden Befehl in der HBase-Shell aus:
hbase(main):001:0> description 'mdds'Table mdds is ENABLEDmddsCOLUMN FAMILIES DESCRIPTION{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1' , VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'FOREVER', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'FOREVER', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}2 Zeile(n) in 0,3440 Sekunden Gescannte Bilder mit Tesseract verarbeiten
OCR hat im Umgang mit Schriftvariationen, Bildrauschen und Ausrichtungsproblemen einen langen Weg zurückgelegt. Hier verwenden wir die Open-Source-OCR-Engine Tesseract, die ursprünglich als proprietäre Software in HP Labs entwickelt wurde. Die Tesseract-Entwicklung wurde seitdem als Open-Source-Software veröffentlicht und seit 2006 von Google gesponsert.
Tesseract ist eine hochportable Softwarebibliothek. Es verwendet die Bildverarbeitungsbibliothek von Leptonica, um ein binäres Bild zu erzeugen, indem es adaptive Schwellenwerte für ein graues oder farbiges Bild durchführt.
Die Verarbeitung folgt einer traditionellen schrittweisen Pipeline. Im Folgenden ist der grobe Ablauf der Schritte aufgeführt:
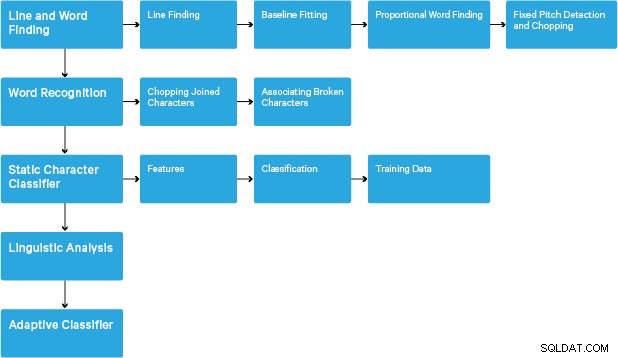
Die Bearbeitung beginnt mit einer angeschlossenen Bauteilanalyse, die zur Speicherung der gefundenen Bauteile führt. Dieser Schritt hilft bei der Überprüfung der Verschachtelung von Umrissen und der Anzahl der untergeordneten und untergeordneten Umrisse.
In diesem Stadium werden Umrisse rein durch Verschachtelung zu Binary Large Objects (BLOBs) zusammengefasst. BLOBs werden in Textzeilen organisiert, und die Zeilen und Bereiche werden auf festen Abstand oder proportionalen Text analysiert. Textzeilen werden je nach Art des Zeichenabstands unterschiedlich in Wörter aufgeteilt. Text mit fester Tonhöhe wird sofort durch Zeichenzellen zerhackt. Proportionaler Text wird mit eindeutigen Leerzeichen und Fuzzy-Leerzeichen in Wörter aufgeteilt.
Die Anerkennung erfolgt dann in einem zweistufigen Verfahren. Im ersten Durchgang wird versucht, jedes Wort der Reihe nach zu erkennen. Jedes zufriedenstellende Wort wird als Trainingsdaten an einen adaptiven Klassifikator weitergegeben. Der adaptive Klassifikator erhält dann die Möglichkeit, Text weiter unten auf der Seite genauer zu erkennen. Da der adaptive Klassifikator möglicherweise zu spät etwas Nützliches gelernt hat, um einen Beitrag am oberen Rand der Seite zu leisten, wird ein zweiter Durchlauf über die Seite gefahren, in dem Wörter, die nicht gut genug erkannt wurden, erneut erkannt werden. Eine abschließende Phase löst unscharfe Leerzeichen auf und prüft alternative Hypothesen für die x-Höhe, um Text in Kapitälchen zu lokalisieren.
Tesseract ist in seiner jetzigen Form voll Unicode-fähig und für mehrere Sprachen trainiert. Basierend auf unseren Recherchen ist es eine der genauesten Open-Source-Bibliotheken, die für OCR verfügbar sind. Wie bereits erwähnt, verwendet Tesseract Leptonica. Wir verwenden auch Ghostscript, um die PDF-Dateien in Bilder aufzuteilen. (Sie können in ein Bildkomprimierungsformat Ihrer Wahl aufteilen; wir haben PNG gewählt.) Diese drei Bibliotheken sind in C++ geschrieben, und um sie von Java/Scala-Programmen aufzurufen, müssen wir Implementierungen entsprechender Java Native Interfaces verwenden. In unserer Arbeit verwenden wir die JNI-Bindings von JavaPresets. (Die Build-Anweisungen finden Sie unten.) Wir haben Scala verwendet, um den Spark-Treiber zu schreiben.
val renderer :SimpleRenderer =neu SimpleRenderer( )renderer.setResolution( 300 )val images:List[Image] =renderer.render( document )
Leptonica liest die geteilten Bilder aus dem vorherigen Schritt ein.
ImageIO.write( x.asInstanceOf[RenderedImage], "png", imageByteStream)val pix:PIX =pixReadMem ( ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ), ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacity())
Wir verwenden dann Tesseract-API-Aufrufe, um den Text zu extrahieren. Wir gehen davon aus, dass die Dokumente hier auf Englisch sind, daher ist der zweite Parameter der Init-Methode „eng.“
val api:TessBaseAPI =new TessBaseAPI( )api.Init( null, "eng" )api.SetImage(pix)api.GetUTF8Text().getString()
Nachdem die Bilder verarbeitet wurden, extrahieren wir einige Felder aus dem Text und senden sie an HBase.
def populateHbase ( fileName:String, lines:String, pdf:org.apache.spark.input.PortableDataStream) :Unit ={ /** HBase-Verbindung konfigurieren und öffnen */ val mddsTbl =_conn.getTable( TableName. valueOf( "mdds" )); val cf ="info" val put =new Put( Bytes.toBytes( fileName )) /** * Felder hier mit Regexes extrahieren * Put-Objekte erstellen und an HBase senden */ val aAndCP ="""(?s)(? m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe:(\w\d\d\d\d\d\d).*"" ".r …….. Zeilen passen { case aAndCP(adr, casenum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "submitter_info" ),Bytes.toBytes( addr ) ).add( Bytes .toBytes( cf ),Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum )) case _ => println( "entsprach keiner Regex" ) } ……. lines.split("\n").foreach { val regNumRegex ="""Regulierungsnummer:\s+(.+)""".r val regNameRegex ="""Regulierungsname:\s+(.+)""" .r …….. ……. _ match { case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "reg_num" ), ……. ….. case _ => print( "" ) } } put.add ( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( Zeilen )) val pdfBytes =pdf.toArray.clone put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( " pdf" ), pdfBytes ) mddsTbl.put( put ) …….} Wenn Sie sich den obigen Code genau ansehen, fügen wir unmittelbar bevor wir das Put-Objekt an HBase senden, die rohen PDF-Bytes in die „obj“-Spaltenfamilie der Tabelle ein. Wir verwenden HBase als Speicherschicht für die extrahierten Felder sowie das Rohbild. Dadurch kann die Anwendung das Originalbild bei Bedarf schnell und bequem extrahieren. Den vollständigen Code finden Sie hier. (Es ist erwähnenswert, dass wir zwar standardmäßige HBase-APIs verwendet haben, um Put-Objekte für HBase zu erstellen, es in einem echten Produktionssystem jedoch ratsam wäre, die Verwendung von SparkOnHBase-APIs in Betracht zu ziehen, die Stapelaktualisierungen von Spark-RDDs auf HBase ermöglichen.)
Ausführungspipeline
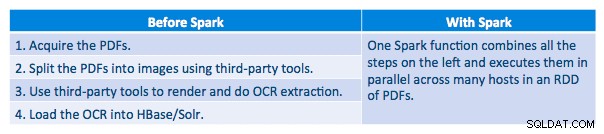
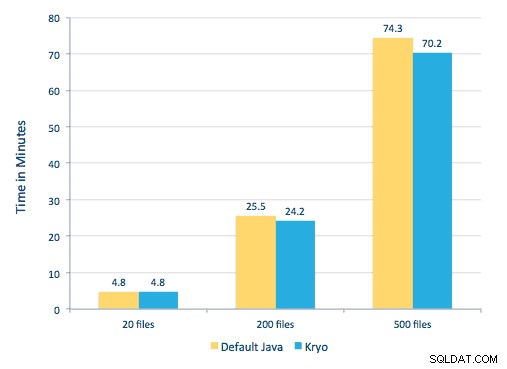
Wir konnten jedes PDF in einem seriellen Rahmen verarbeiten. Um die Verarbeitung zu skalieren, haben wir uns entschieden, diese PDFs auf verteilte Weise mit Spark zu verarbeiten. Das folgende Diagramm zeigt, wie wir verschiedene Phasen dieser Verarbeitung kombinieren, um den Workflow in einen einfachen Makroaufruf von Spark zu verwandeln und die Daten in HBase zu laden.
Wir haben auch versucht, einen Vergleich zwischen Serialisierungsmethoden anzustellen, aber mit unserem Datensatz konnten wir keinen signifikanten Leistungsunterschied feststellen.
Umgebungseinrichtung
Verwendete Hardware:Fünf-Knoten-Cluster mit 15 GB Arbeitsspeicher, 4 vCPUs und 2 x 40 GB SSD
Da wir C++-Bibliotheken für die Verarbeitung verwendet haben, haben wir die JNI-Bindungen verwendet, die hier zu finden sind.
Erstellen Sie die JNI-Bindungen für Tesseract und Leptonica aus javaCPP-Voreinstellungen:
-
- Auf allen Knoten:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel git clone https://github.com/bytedeco/javacpp-presets.gitcd javacpp-presets- Leptonica erstellen.
cd leptonica./cppbuild.sh install leptonicad cppbuild/linux-x86_64/leptonica-1.72/LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&sudo make installcd ../../../mvn clean installcd ..
- Baue Tesserakt.
- Auf allen Knoten:
cd tesseract./cppbuild.sh install tesseractcd tesseract/cppbuild/linux-x86_64/tesseract-3.03LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&make installcd ../ ../../mvn clean installcd ..
- JavaCPP-Voreinstellungen erstellen.
mvn clean install --projects leptonica,tesseract
Wir verwenden Ghostscript, um die Bilder aus den PDFs zu extrahieren. Anweisungen zum Erstellen von Ghostscript, die den hier verwendeten Versionen von Tesseract und Leptonica entsprechen, lauten wie folgt. (Stellen Sie sicher, dass Ghostscript nicht über den Paketmanager im System installiert ist.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gztar zxvf ghostscript-9.16.tar.gzcd ghostscript-9.16./autogen.sh &&./configure --prefix=/usr - -disable-compile-inits --enable-dynamicsudo make &&make soinstall &&install -v -m644 base/*.h /usr/include/ghostscript &&ln -v -s ghostscript /usr/include/ps(Abhängig von Ihrem ldpath Einstellung, die Sie möglicherweise vornehmen müssen):sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Stellen Sie sicher, dass sich alle benötigten Bibliotheken im Klassenpfad befinden. Wir legen alle relevanten JAR-Dateien in einem Verzeichnis namens lib ab. Komma ist unten wichtig:
$ für i in `ls lib/*`; exportiere MY_JARS=./$i,$MY_JARS; donetesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Wir rufen das Spark-Programm wie folgt auf. Wir müssen den extraLibraryPath für native Ghostscript-Bibliotheken angeben; die andere conf wird für Tesseract benötigt.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor-cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --confspark.executorEnv. TESSDATA_PREFIX=/home/vsingh/javacpp-presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --confspark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase /lib/htrace-core-3.1.0-incubating.jar --driver-class-path/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0 -incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer--conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Erstellen einer Solr-Sammlung
Solr lässt sich über Lily HBase Indexer ziemlich nahtlos in HBase integrieren. Um zu verstehen, wie die Integration der Lily Indexer-Integration mit HBase erfolgt, können Sie unseren vorherigen Beitrag im Abschnitt „HBase-Replikation und Lily HBase Indexer verstehen“ auffrischen.
Nachfolgend skizzieren wir die Schritte, die zum Erstellen der Indizes durchgeführt werden müssen:
- Generieren Sie eine schema.xml-Beispielkonfigurationsdatei:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg - Bearbeiten Sie die Datei schema.xml in
$HOME/solrcfg, indem Sie die Felder angeben, die wir für unsere Sammlung benötigen. Die vollständige Datei finden Sie hier. - Laden Sie die Solr-Konfigurationen in ZooKeeper hoch:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - Generieren Sie die Solr-Sammlung mit 2 Shards (-s 2) und 2 Replicas (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
Im obigen Befehl haben wir eine Solr-Sammlung mit zwei Shards (-s 2) und zwei Replikaten (-r 2) Parametern erstellt. Die Parameter waren für unseren Korpus ausreichend, aber in einer tatsächlichen Bereitstellung müsste man die Zahl basierend auf anderen Überlegungen festlegen, die außerhalb unseres Diskussionsbereichs hier liegen.
Registrieren des Indexers
Dieser Schritt ist erforderlich, um den Indexer und die HBase-Replikation hinzuzufügen und zu konfigurieren. Der folgende Befehl aktualisiert ZooKeeper und fügt mdds_indexer als Replikationspeer für HBase hinzu. Es fügt auch Konfigurationen in ZooKeeper ein, die Lily HBase Indexer verwendet, um auf die richtige Sammlung in Solr zu verweisen. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argumente:
-n mdds_indexer– gibt den Namen des Indexers an, der in ZooKeeper registriert wird-c indexer-config.xml– Konfigurationsdatei, die das Verhalten des Indexers festlegt-cp solr.zk=localhost:2181/solr– gibt den Speicherort von ZooKeeper und Solr config an. Dies sollte mit dem umgebungsspezifischen Standort von ZooKeeper aktualisiert werden.-cp solr.collection=mdds_collection– gibt an, welche Sammlung aktualisiert werden soll. Erinnern Sie sich an den Solr-Konfigurationsschritt, in dem wir collection1 erstellt haben.
Die index-config.xml Datei ist in diesem Fall relativ einfach; alles, was es tut, ist, dem Indexer anzugeben, welche Tabelle betrachtet werden soll, die Klasse, die als Mapper verwendet wird (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ) und den Speicherort der Morphline-Konfigurationsdatei. Standardmäßig ist der Mapping-Typ auf row gesetzt , in diesem Fall wird das Solr-Dokument zur vollständigen Zeile. Param name="morphlineFile" gibt den Speicherort der Morphlines-Konfigurationsdatei an. Der Speicherort könnte ein absoluter Pfad Ihrer Morphlines-Datei sein, aber da Sie Cloudera Manager verwenden, geben Sie den relativen Pfad als morphlines.conf an.
Den Inhalt der hbase-indexer Konfigurationsdatei finden Sie hier.
Konfigurieren und Starten von Lily HBase Indexer
Wenn Sie Lily HBase Indexer aktivieren, müssen Sie die Morphlines-Transformationslogik spezifizieren, die es diesem Indexer ermöglicht, Aktualisierungen der Medizingerätetabelle zu parsen und alle relevanten Felder zu extrahieren. Gehen Sie zu Dienste und wählen Sie Lily HBase Indexer, den Sie zuvor hinzugefügt haben. Wählen Sie Konfigurationen->Anzeigen und Bearbeiten->Dienstweit->Morphlines aus . Kopieren Sie die Morphlines-Datei und fügen Sie sie ein.
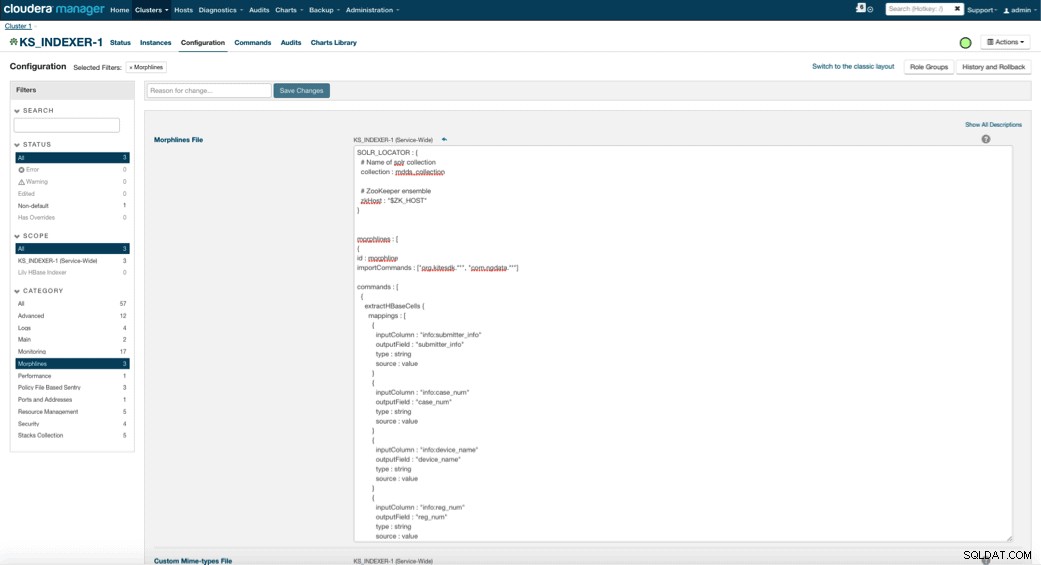
Die Morphline-Bibliothek für medizinische Geräte führt die folgenden Aktionen aus:
- Lesen Sie die HBase-E-Mail-Ereignisse mit
extractHBaseCellsBefehl - Konvertieren Sie die Datums-/Zeitstempel in ein Feld, das Solr versteht, mit dem
convertTimestampBefehle - Löschen Sie alle zusätzlichen Felder, die wir nicht in schema.xml angegeben haben, mit
sanitizeUknownSolrFieldsBefehl
Laden Sie hier eine Kopie dieser Morphlines-Datei herunter.
Ein wichtiger Hinweis ist, dass das ID-Feld automatisch von Lily HBase Indexer generiert wird. Diese Einstellung kann in der Datei index-config.xml oben konfiguriert werden, indem das Attribut unique-key-field angegeben wird. Es empfiehlt sich, den Standardnamen id beizubehalten – da er in der obigen XML-Datei nicht angegeben wurde, wurde das Standard-ID-Feld generiert und ist eine Kombination aus RowID.
Zugriff auf die Daten
Sie haben die Wahl zwischen vielen visuellen Tools, um auf die indizierten Bilder zuzugreifen. HUE und Solr GUI sind beide sehr gute Optionen. HBase ermöglicht auch eine Reihe von Zugriffstechniken, nicht nur über eine GUI, sondern auch über die HBase-Shell, API und sogar einfache Skripttechniken.
Die Integration mit Solr gibt Ihnen große Flexibilität und kann auch sehr einfache sowie erweiterte Suchoptionen für Ihre Daten bereitstellen. Wenn Sie beispielsweise die Solr-Datei schema.xml so konfigurieren, dass alle Felder innerhalb des E-Mail-Objekts in Solr gespeichert werden, können Benutzer über eine einfache Suche auf den vollständigen Nachrichtentext zugreifen, wobei Speicherplatz und Rechenkomplexität in Kauf genommen werden müssen. Alternativ können Sie Solr so konfigurieren, dass nur eine begrenzte Anzahl von Feldern gespeichert wird, z. B. die ID. Mit diesen Elementen können Benutzer Solr schnell durchsuchen und die Zeilen-ID abrufen, die wiederum verwendet werden kann, um einzelne Felder oder das gesamte Bild aus HBase selbst abzurufen.
Das obige Beispiel speichert nur die rowID in Solr, indiziert jedoch alle aus dem Bild extrahierten Felder. Beim Durchsuchen von Solr in diesem Szenario werden HBase-Zeilen-IDs abgerufen, die Sie dann zum Abfragen von HBase verwenden können. Diese Art der Einrichtung ist ideal für Solr, da sie die Speicherkosten niedrig hält und die Indizierungsfunktionen von Solr voll ausschöpft.
Beispielabfragen
Nachfolgend finden Sie einige Beispielabfragen, die von der Anwendung in Solr ausgeführt werden können. Die Idee ist, dass der Client zunächst Solr-Indizes abfragt und die RowID von HBase zurückgibt. Fragen Sie dann HBase nach den restlichen Feldern und/oder dem ursprünglichen Rohbild ab.
- Geben Sie mir alle Dokumente, die zwischen den folgenden Daten eingereicht wurden:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01 -06T23:59:59.999Z BIS 2010-02-06T23:59:59.999Z]
- Geben Sie mir Dokumente, die unter dem Zulassungsnamen für mobile Röntgensysteme abgelegt wurden:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile Röntgensystem
- Geben Sie mir alle Dokumente, die von chinesischen Herstellern eingereicht wurden:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
Die IDs aus Solr-Dokumenten sind die Zeilen-IDs in HBase; der zweite Teil der Abfrage geht an HBase, um die Daten zu extrahieren (einschließlich des Roh-PDF, falls erforderlich).
Zugriff über HUE
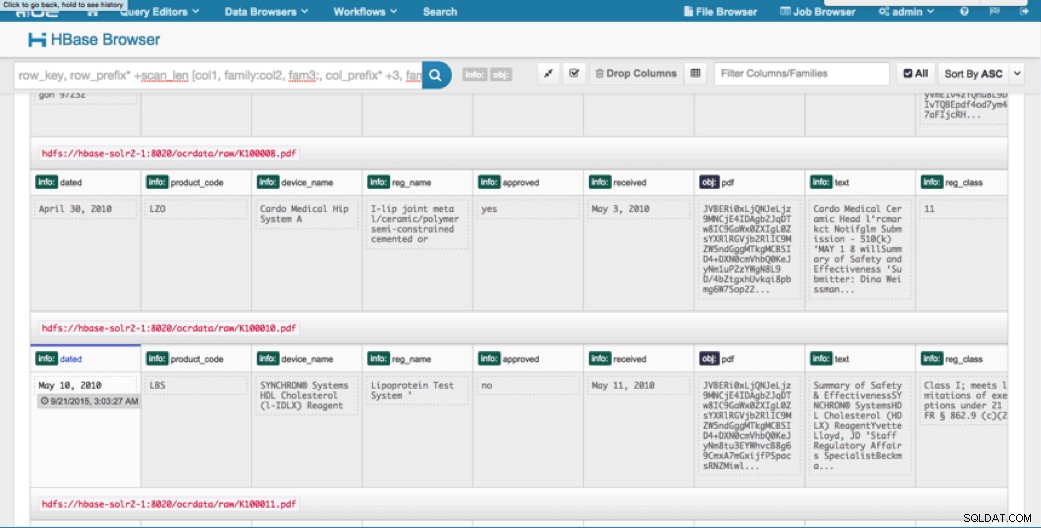
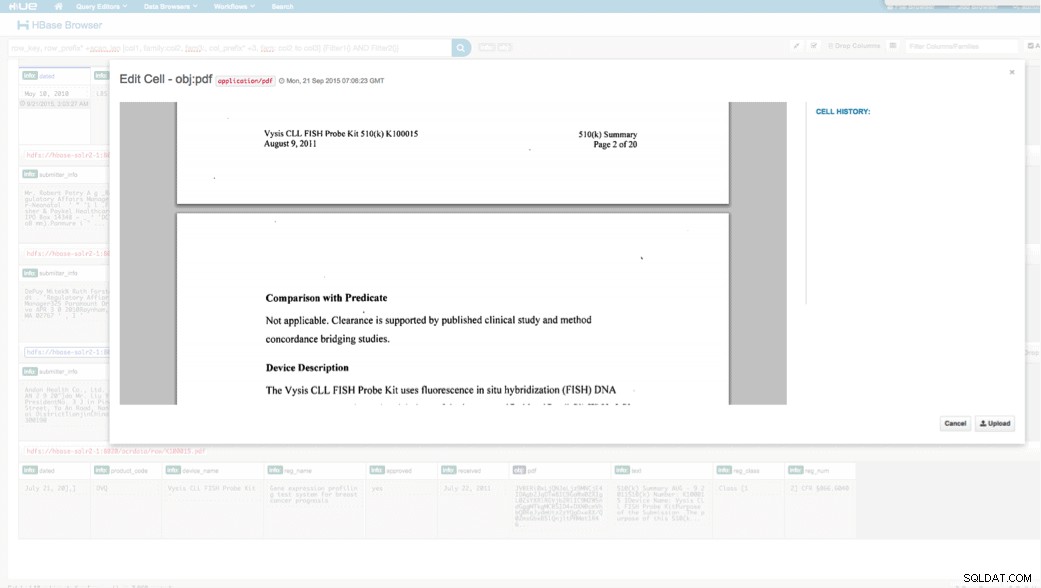
Wir können die hochgeladenen Daten über den HBase-Browser in HUE anzeigen. Eine großartige Sache an HUE ist, dass es die Binärdateien für PDF erkennen und sie rendern kann, wenn darauf geklickt wird.
Unten sehen Sie eine Momentaufnahme der Ansicht der geparsten Felder in HBase-Zeilen sowie eine gerenderte Ansicht eines der PDF-Objekte, die als MOB in der obj-Spaltenfamilie gespeichert sind.
Schlussfolgerung
In diesem Beitrag haben wir gezeigt, wie man Standard-Open-Source-Technologien verwendet, um OCR auf gescannte Dokumente mit einem skalierbaren Spark-Programm durchzuführen, in HBase für einen schnellen Abruf zu speichern und die extrahierten Informationen in Solr zu indizieren. Es sollte offensichtlich sein, dass:
- Angesichts des Nachrichtenspezifikationsformats können wir Felder und Wertepaare extrahieren und sie über Solr durchsuchbar machen.
- Diese Felder aus Daten können die IDMP-Anforderungen erfüllen, die Legacy-Daten elektronisch zu machen, die irgendwann im nächsten Jahr in Kraft treten.
- Die Felder sowie Rohbilder können in HBase gespeichert und über Standard-APIs abgerufen werden.
Wenn Sie gescannte Dokumente verarbeiten und die Daten mit verschiedenen anderen Quellen in Ihrem Unternehmen kombinieren müssen, sollten Sie eine Kombination aus Spark, HBase, Solr zusammen mit Tesseract und Leptonica in Betracht ziehen. Es kann Ihnen viel Zeit und Geld sparen!
Jeff Shmain ist Senior Solution Architect bei Cloudera. Er verfügt über mehr als 16 Jahre Erfahrung in der Finanzbranche mit ausgeprägtem Verständnis für Wertpapierhandel, Risiken und Vorschriften. In den letzten Jahren hat er bei 8 der 10 weltweit größten Investmentbanken an verschiedenen Use-Case-Implementierungen gearbeitet.
Vartika Singh ist Senior Solution Consultant bei Cloudera. Sie hat über 12 Jahre Erfahrung in angewandtem maschinellem Lernen und Softwareentwicklung.